FastKing gDNA Dispelling RT SuperMix
Funktioner
■ Snabbt: Ett steg för att slutföra genomavlägsnande och effektiv omvänd transkription inom 18 minuter genom att bara lägga till mallar.
■ Hög effektivitet: Omvänt transkriptas modifieras med ett hydrofobt motiv, med RT -effektivitet över 95%.
■ Enkelt och enkelt: Den exklusiva värmekänsliga DNasen har snabb effekt, hög effektivitet med kortare reaktionstid och påverkar inte cDNA.
Specifikation
Typ: Genmodifierat omvänt transkriptas, gDNas
Förfaranden: Ett steg (genomiskt DNA-avlägsnande och RT)
RT -effektivitet: > 95%
Mall: 1 ng- 2 μg
Driftstid: ~ 18 min
Ansökningar: Det omvända transkriberade cDNA kan användas i konventionell PCR, realtids -PCR, cDNA -bibliotekskonstruktion, SAGE (Serial Analysis of Gene Expression), primerförlängning och andra konventionella experiment.
Alla produkter kan anpassas för ODM/OEM. För detaljer,vänligen klicka på Anpassad tjänst (ODM/OEM)
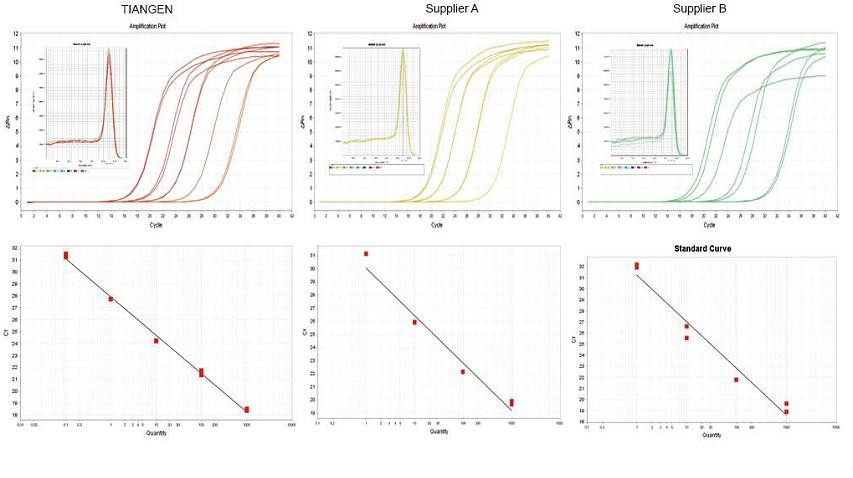 |
Experimentellt exempel 1. cDNA syntetiserades med användning av ettstegs omvända kvantitativa reagens från TIANGEN FastKing gDNA Dispelling RT SuperMix, relevanta produkter från leverantör A respektive leverantör B. Upptäck RN5 -genen hos möss med TIANGEN Talent qPCR PreMix (SYBR Green), och amplifieringskurvan, smältkurvan och standardkurvan analyserades. Resultaten visar att TIANGEN FastKing gDNA Dispelling RT SuperMix har det högsta kvantitativa Ct -värdet efter omvänd transkription och utmärkt spänningsmotstånd, och har uppenbara fördelar för mallar med höga orenhetsrester. |
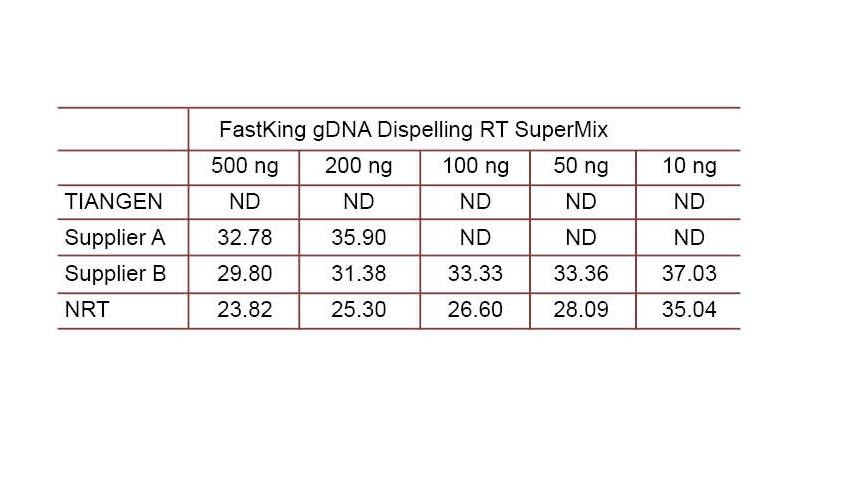 |
Experimentellt exempel 2. cDNA syntetiserades med användning av ettstegs omvända kvantitativa reagens från TIANGEN FastKing gDNA Dispelling RT SuperMix, relevanta produkter från leverantör A respektive leverantör B. Upptäck mänsklig HsG -gen med TIANGEN Talent qPCR PreMix (SYBR Green) och lägg manuellt till olika koncentrationer av genomiskt DNA för att upptäcka förmågan att avlägsna gDNA för olika reagens. Ct -resultat visar att TIANGEN FastKing gDNA Dispelling RT SuperMix har utmärkt förmåga att ta bort genomiskt DNA. Upp till 500 ng genomisk DNA -rest kan avlägsnas perfekt utan att påverka resultaten. ND: Inte upptäckt. NRT: Detektering av blandningen utan omvänd transkription. |
A-1 RNA bryts ned
—— Rena högkvalitativt RNA utan förorening. Materialet från vilket RNA extraheras bör vara så färskt som möjligt för att förhindra nedbrytning av RNA. Analysera RNA -integritet på denaturerad gel före RT -reaktion. Efter RNA -extraktion bör den förvaras i 100% formamid. Om RNas -hämmare används bör uppvärmningstemperaturen vara <45 ° C och pH bör vara lägre än 8,0, annars släpper hämmaren allt bundet RNas. Dessutom bör RNas -hämmare tillsättas i lösningar som innehåller ≥ 0,8 mM DTT.
A-2 RNA innehåller hämmare av omvända transkriptionsreaktioner
—— Omvända transkriptionshämmare inkluderar SDS, EDTA, glycerol, natriumpyrofosfat, spermidin, formamid, guanidinsalt, etc. Blanda kontroll -RNA med provet och jämför utbytet med kontroll -RNA -reaktionen för att kontrollera om det finns en hämmare. Tvätta RNA -utfällning med 70% (volym/volym) etanol för att avlägsna hämmare.
A-3 Otillräcklig glödgning av primrar som används för syntetisering av den första strängen av cDNA
—— Bestäm att glödgningstemperaturen är lämplig för de primrar som används i experimentet. För slumpmässiga hexamerer rekommenderas att temperaturen hålls vid 25 ° C i 10 minuter innan reaktionstemperaturen uppnås. För genspecifika primers (GSP), försök med annan GSP, eller byt till oligo (dT) eller slumpmässig hexamer.
A-4 Liten mängd start-RNA
—— Öka mängden RNA. För RNA -prover mindre än 50 ng kan 0,1 μg till 0,5 μg acetyl BSA användas vid den första strängens cDNA -syntes
A-5 Målsekvensen uttrycks inte i de analyserade vävnaderna.
—— Testa andra vävnader.
A-6 PCR-reaktion misslyckas
—— För tvåstegs RT-PCR kan cDNA-mallen i PCR-steget inte överstiga 1/5 av reaktionsvolymen.
A-1 Ospecifik glödgning av primers och mallar
—— 3'-änden av primers bör inte innehålla 2-3 dG eller dC. Använd Genspecifika primers i den första strängssyntesen istället för slumpmässiga primrar eller oligo (dT). Använd en högre glödgningstemperatur under de första cyklerna och sedan en lägre glödgningstemperatur. Använd varmstart Taq DNA-polymeras för PCR för att förbättra specificiteten av reaktionen.
A-2 Dålig design av genspecifika primers
——Följ samma principer för förstärkning av primer.
A-3 RNA förorenat med genomiskt DNA
——Behandla RNA med DNas av PCR-klass I. Skapa en kontrollreaktion utan omvänd transkription för att upptäcka DNA-kontaminering.
A-4 Formning av primer dimer
—— Designa primers utan komplementära sekvenser i 3' -änden.
A-5 För hög mg2+ koncentration
——Optimera Mg2+ koncentration för varje mall och primerkombination
A-6 Förorenat med främmande DNA
—— Använd aerosolresistenta spetsar och UDG-enzymer.
A-1 Innehållet i den första strängprodukten är för högt
—— Minska mängden av den första strängprodukten i det konventionella PCR -reaktionssteget.
A-2 För hög grundmängd i PCR-reaktion
—— Minska primerinmatning.
A-3 För många cykler
—— Optimera PCR -reaktionsförhållandena och minska antalet PCR -cykler.
A-4 För låg glödgningstemperatur
—— Öka glödgningstemperaturen för att förhindra ospecifik initiering och förlängning.
A-5 Ospecifik amplifiering av oligonukleotidfragment som genereras av DNas nedbrytning av DNA —— Extrahera högkvalitativt RNA för att förhindra DNA-kontaminering.
RT-PCR är att reversera transkribera RNA till cDNA och sedan använda det omvända transkriberade cDNA som en mall för PCR-reaktion för att förstärka målfragmentet. Välj antingen slumpmässiga primrar, Oligo dT och genspecifika primrar enligt experimentets specifika förhållanden. Alla ovanstående primrar kan användas för kort eukaryot cell -mRNA utan hårnålsstruktur.
Slumpmässig primer: Lämplig för långt RNA med hårnålsstruktur, liksom alla typer av RNA som rRNA, mRNA, tRNA, etc. De används huvudsakligen för RT-PCR-reaktion av en enda mall.
Oligo dT: Lämplig för RNA med PolyA -tailing (prokaryot RNA, eukaryot Oligo dT rRNA och tRNA har inte PolyA -svansar). Eftersom Oligo dT är bunden till PolyA-svans måste kvaliteten på RNA-prover vara hög, och även en liten mängd nedbrytning kommer att kraftigt minska mängden cDNA-syntes i full längd.
Genspecifik primer: Komplementär till mallsekvensen, lämplig för situationer där målsekvensen är känd.
Det finns två sätt:
1. Intern referensmetod: I teorin är cDNA DNA -fragment av olika längd, så resultatet av elektrofores är utstryk. Om RNA -mängden är låg kommer ingen produkt att visas vid elektrofores, men det betyder inte att ingen produkt kommer att förstärkas med PCR. I allmänhet kan intern referens användas för att detektera cDNA. Om den interna referensen har resultat kan kvaliteten på cDNA i princip garanteras (i några få fall, om målgenfragmentet är för långt, kan det finnas undantag).
2. Om det finns en känd gen som förstärks med denna mall, kan den verifieras av primrarna för denna gen. Förstärkningen av intern referens betyder inte nödvändigtvis att det inte finns några problem med cDNA. Eftersom intern referens har ett stort överflöd av cDNA är det lätt att amplifiera. Om cDNA försämras delvis av olika anledningar, ur sannolikhetsperspektivet, kommer PCR -resultat av låggenoms målgener att påverkas kraftigt. Medan intern referens fortfarande är hög i överflöd, kommer amplifieringen sannolikt inte att påverkas.
Delvis nedbrytning av RNA. Upptäck integriteten och rena RNA
RNA -innehållet i olika arter kan vara olika, men i allmänhet bör det extraherade totala RNA innehålla två klara 28S- och 18S -band i gelelektrofores, och ljusstyrkan för det tidigare bandet bör vara dubbelt så hög som den för det senare. 5S -bandet indikerar att RNA har försämrats och dess ljusstyrka är proportionell mot graden av nedbrytning. Den framgångsrika förstärkningen av intern referens betyder inte att det inte finns några problem med RNA, eftersom den interna referensen är i överflöd kan RNA amplifieras så länge nedbrytningen inte är allvarlig. OD260/OD280förhållandet mellan rent RNA mätt med spektrofotometer bör vara mellan 1,9 och 2,1. En liten mängd proteinrenhet i RNA kommer att minska förhållandet. Så länge värdet inte är för lågt påverkas inte RT. Det viktigaste för RT är RNA -integritet.
Förlängningen av den interna referensgenen kan bara indikera att RT har lyckats, men det är inte nödvändigtvis relaterat till kvaliteten på cDNA -strängen. Eftersom de interna referensfragmenten i allmänhet är små i storlek och höga i uttryck, är de lättare att lyckas med omvänd transkription. Storleken och uttrycket för målgenen varierar emellertid från gen till gen. CDNA -kvaliteten kan inte bedömas endast genom intern referens speciellt för målfragmenten längre än 2 kb.
Vissa prover har komplexa sekundära strukturer, eller har ett rikt GC -innehåll, eller är värdefulla med låga mängder. I dessa fall bör lämpligt omvänt transkriptas väljas utifrån målfragmentets storlek och provet. För RNA -mallar med högt GC -innehåll och komplex sekundärstruktur är det svårt att öppna den sekundära strukturen vid låg temperatur eller med vanligt omvänt transkriptas. För dessa mallar kan Quant Reverse Transkriptas väljas, eftersom dess omvända transkriptionsprestanda uppenbarligen är bättre än M-MLV-seriens omvänt transkriptas, vilket kan reversera transkribera olika RNA-mallar effektivt och transkribera RNA till cDNA första sträng i maximal utsträckning. När du använder ett generellt omvänt transkriptas -kit kan 20 μl -systemet endast effektivt reversera transkribera 1 μg totalt RNA. Var uppmärksam på kitets maximala RT -kapacitet. Om mallen läggs till i överskott kommer omvänd transkription att gynna RNA med hög överflöd. Därför är det bättre att inte överskrida systemets maximala kapacitet.
A-1 Avgör om RNA bryts ned kraftigt och om RT är framgångsrikt
I allmänhet orsakas orsaken till misslyckande av intern referensförstärkning ofta av allvarlig RNA -nedbrytning. En annan möjlig orsak är omvänd transkriptionsfel. Internreferens kan inte användas som en standard för att bedöma kvaliteten på cDNA -enkelsträng, men den kan användas som en standard för att bedöma om omvänd transkription är framgångsrik om det inte finns några problem med RNA -kvaliteten. Det viktigaste i omvänd transkription är att upprätthålla en konstant temperatur och ett konstant reaktionssystem för att förbättra reaktionseffektiviteten.
A-2 Avgör om primrarna för amplifiering av interna referensgener är tillförlitliga och om det finns några problem med reagenser som används i PCR.
För relativ kvantifiering måste RNA kvantifieras före omvänd transkription, vilket också krävs i många omvända transkriptionssatser, till exempel kvantifiera RNA -ingången som 1 μg. Eftersom det omvända transkriberade cDNA är en blandad lösning, inklusive RNA, oligo dT, enzym, dNTP och till och med lite DNA -rester, kommer avvikelse att orsakas, så det är omöjligt att exakt kvantifiera cDNA. Därför är RNA -kvantifiering nödvändig. Med tanke på den omvända transkriptionseffektiviteten är densamma bland olika prover, bör mängden cDNA som erhålls vara densamma, och den kvantitativa analysen kan visa jämförelse av expressionsnivåer för olika gener i samma mängd totalt RNA. När kvantitativ PCR för relativ fluorescens utförs kan det hända att kvantitativt cDNA inte krävs efter omvänd transkription eftersom den interna referensgenen kan fungera som referens.
Det är främst relaterat till generna, och omvänd transkription av långt fragment är inte genomförbart för de flesta gener. För det första är effektiviteten av omvänd transkription mycket lägre än för PCR. För det andra begränsar den GC -rika regionen och sekundära strukturen hos många gener både omvänd transkription och PCR. Slutligen är trovärdigheten och förstärkningseffektiviteten hos PCR svår att garantera samtidigt. I processen med omvänd transkription kan ingen garantera att få långt fragment för gener med låg kopia, särskilt med hjälp av oligo dT. När det gäller 5 'UTR med mer GC är det ännu svårare. Därför är det fortfarande en rimlig metod att vända transkript med slumpmässiga primers, hitta de naturliga klyvningsställena i målfragmentet, förstärka med segment och sedan utföra restriktionssmältningen och ligeringen. I allmänhet är det svårt att direkt amplifiera fragment större än 2 kb, men det är inte alltid omöjligt att erhålla: 1. Först och främst garantera integriteten för RNA/mRNA, och TRIZOL -extraktion är att föredra. 2.M-MLV RT-PCR-kit kan användas direkt. Förläng glödgningstiden och öka cykelantalet i förstärkningsprocessen ordentligt. Alternativt kan kapslad PCR appliceras eller utföra en eller två reaktioner först med lämpligt förlängd denaturering och förlängningstid före normal PCR -förstärkning, vilket kan hjälpa till att förlänga fragment. Var uppmärksam på polymeras trohet. 3. Long Taq kan användas i PCR för att få perfekta resultat. 4.För proteinuttryckstillämpning bör högfrekvent polymeras appliceras.
Det finns två typer av omvänt transkriptas som erbjuds av TIANGEN: Quant/King RTase och TIANScript M-MLV. Den största skillnaden mellan dem är inmatningsmängden mallar. Quant är ett unikt omvänt transkriptas, som skiljer sig från det vanliga M-MLV som härrör från Moloney murin leukemivirus. Quant är ett nytt högeffektivt omvänt transkriptas som rekombinant uttrycks av Escherichia coli. Quant är lämplig för amplifiering av 50 ng-2 μg RNA med hög omvänd transkriptionell aktivitet och högt utbyte. Jämfört med vanliga MMLV eller AMV är Quants största egenskap att den har mycket stark affinitet med RNA -mallar och kan vända transkriberade komplexa mallar utan högtemperatur -denaturering. För mallar med högre GC -innehåll är omvänd effektivitet högre. Detta omvänt transkriptas har emellertid RNas H -aktivitet, vilket kan påverka cDNA -produktens längd (lämplig för <4,5 kb mallar). För konventionell omvänd transkription rekommenderas TIANScript MMLV omvänd transkriptas. Detta RTas är ett modifierat enzym med mycket svag RNas H -aktivitet, vilket är lämpligt för lång (> 5 kb) cDNA -syntes.
Ett-stegs omvänd transkription och PCR-amplifiering slutförs i samma rör utan att öppna rörlocket mellan cDNA-syntes och amplifiering, vilket är till hjälp för att minska kontaminering. Eftersom alla erhållna cDNA -prover används för amplifiering är känsligheten högre, med minst 0,01 pg totalt RNA. För framgångsrik RTPCR i ett steg används generellt specifika primers för att initiera cDNA-syntes. Tvåstegsmetoden, nämligen omvänd transkription och PCR-amplifiering utförs i två steg. För det första utförs omvänd transkription från en RNA -mall för att erhålla cDNA, och det erhållna cDNA utsätts för en eller flera olika PCR -reaktioner. Tvåstegsmetoden kan använda oligo (dT) eller slumpmässiga primrar för att styra syntesen av den första strängen av cDNA, och kan reversera transkribera all mRNA-information från ett specifikt prov.
Produktkategorier
VARFÖR VÄLJA OSS
Sedan etableringen har vår fabrik utvecklat produkter i världsklass med principen
av kvalitet först. Våra produkter har fått ett utmärkt rykte i branschen och är värdefulla bland nya och gamla kunder.